- 首页
- 论文服务
- 科研支持
- 访学通
- 美捷登学院
RNase RRibonuclease R
试剂组分
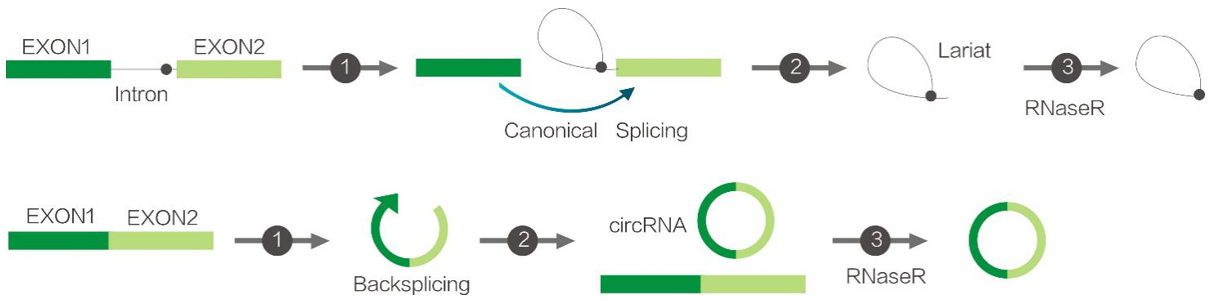
RNase R (Ribonuclease R)是一种来源于大肠杆菌RNR超家族的3’-5’核糖核酸外切酶,可从3’-5’方向将RNA逐步切割成二核苷酸和三核苷酸。RNase R可消化几乎所有的线性RNA分子,但不易消化环形RNA、套索结构或3’突出末端少于7个核苷酸的双链RNA分子。RNase R常用于基因表达和可变剪切研究,可消化线性RNA以使环形RNA (circRNAs)或套索结构RNA (lariat RNA)得到富集。
产品组分:

单位定义:
标准反应体系下,于37℃,10 min将1 μg poly(A)转化成酸溶核苷酸所需的酶量定义为一个活力单位(U)。
反应体系:20 mM Tris-HCl (pH 8.0), 100 mM KCl, 0.1 mM MgCl2, 37℃孵育。
注:1)RNase R在0.1-0.5 mM Mg2+存在时活性最高,反应中EDTA可能降低RNase R活性,此时可额外添加MgCl2使Mg2+浓度最高达到0.5mM。2)孵育后可于70℃,10 min使酶失活,再进行下游实验(如逆转录);也可不灭活,直接纯化后进行下游实验。
质量控制:
SDS-PAGE检测纯度>95%;Total RNA经RNase R消化后进行RT-qPCR检测,线性RNA丰度明显降低,环形RNA丰度基本不变。
RNase R消化实验:
1 反应体系
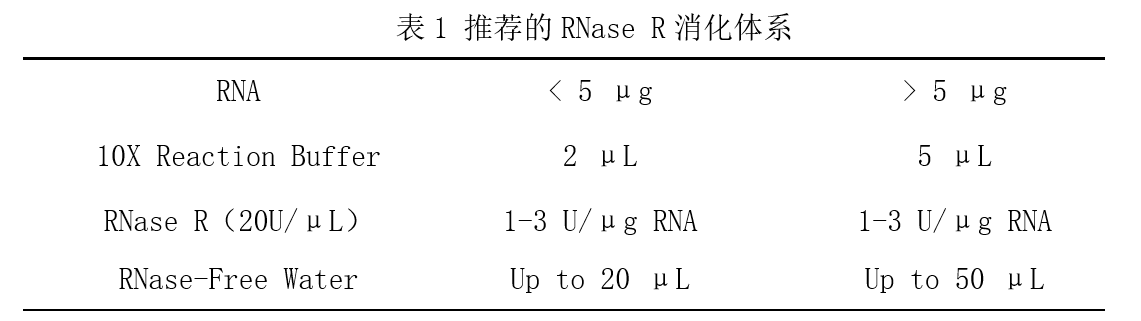
*:RNase R反应体系需根据实验进行调整。一般情况下,3 U RNase R在37℃下反应15min几乎完全消除1μg 线性RNA。
2 纯化回收
消化后的RNA可使用苯酚:氯仿:异戊醇(25: 24:1, V: V)溶液(或Trizol Reagent)抽提,再使用乙醇沉淀回收;或者使用RNA纯化柱或磁珠进行纯化回收。
注:经RNase R消化后可不做纯化,70℃保持10 min失活RNase R后可直接进行RT-PCR。
常见问题:
1 消化时间的选择
一般10-30 min即可消化掉大部分线性RNA,PCR检测线性RNA丰度有几百倍的降低,也可按需求适当延长消化时间,但不推荐1h以上的消化,因为时间过长可能导致少数耐受力弱的circRNAs被消化。
2 内参的选择与定量计算
1)情况1:RNase R消化后“直接”进行RT-qPCR检测对消化组(RNase R+)与对照组(RNase R-)的circRNA的进行定量计算时,应统一以对照组(RNase R-)的β-actin或GAPDH为内参。
2)情况1:RNase R消化后“纯化回收”再进行RT-qPCR检测在纯化前加入少量其他物种的RNA作为外参,统一以外参标准化样品后再进行计算。
3 实验结果一致性(重复性)差
1)RNase R消化/逆转录/PCR过程中加样不准确;
2)RNase R的用量有偏差;
3)其他RNase污染。
4 线性RNA消化不成功
1)反应体系中的NaCl浓度过高抑制了RNase R的活性,或存在RNase R的失活剂EDTA;
2)某些没有3’突出末端的结构化线性RNA或含有G-四联体的线性RNA可能耐受RNase R消化。
5 环状RNA也被消化了
1)外源RNase污染,使用RNase-Free耗材,反应体系可适当加入RNase inhibitor;
2)少数circRNAs耐受RNase R消化力弱的,可尝试减少RNase R用量或缩短消化时间。
- 上一篇:没有了
- 下一篇:没有了
- 返回栏目页
公司地址:武汉市江岸区中山大道岳飞街21号金源世界中心D座1201-1203室
Copyright © 2024 Medjaden All Rights Reserved. 版权所有 不得翻录
 鄂ICP备11002610号-1
鄂公网安备42010202000197
联系电话:027-85567377
鄂ICP备11002610号-1
鄂公网安备42010202000197
联系电话:027-85567377
联系人:陈经理 - 科研支持




 关注美捷登公众号
关注美捷登公众号
 美捷登公益直播
美捷登公益直播




